Package R ggplot2 : Différence entre versions
(Test, just a test) |
|||
| (16 révisions intermédiaires par 3 utilisateurs non affichées) | |||
| Ligne 30 : | Ligne 30 : | ||
head(iris) | head(iris) | ||
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:Headiris.png]] | ||
Attention ! pour utiliser ggplot2, les données doivent être sous la forme « data.frame », ce qui peut être vérifié par : class(dataset), et corrigé si besoin avec as.data.frame(dataset) | Attention ! pour utiliser ggplot2, les données doivent être sous la forme « data.frame », ce qui peut être vérifié par : class(dataset), et corrigé si besoin avec as.data.frame(dataset) | ||
| Ligne 72 : | Ligne 74 : | ||
'''Pour deux variables :''' | '''Pour deux variables :''' | ||
| − | * point : scatterplot | + | * point : scatterplot (deux variables continues) |
| − | * smooth : trace une courbe de tendance et son écart-type | + | * smooth : trace une courbe de tendance et son écart-type (deux variables continues) |
| − | * boxplot : boxplot | + | * boxplot : boxplot (une variable quantitative continue, une variable discrète) |
| + | |||
| + | '''Séries temporelles''' | ||
* line : trace des lignes entre les points (de la gauche vers la droite) | * line : trace des lignes entre les points (de la gauche vers la droite) | ||
* path : trace des lignes entre les points (dans n’importe quelle direction) | * path : trace des lignes entre les points (dans n’importe quelle direction) | ||
| Ligne 91 : | Ligne 95 : | ||
</pre> | </pre> | ||
| − | On peut l'améliorer : changer la couleur de remplissage (argument fill) et/ou la couleur de la bordure (argument colour) : | + | [[Fichier:histo_base_qplot.png]] |
| + | |||
| + | On peut l'améliorer : changer la couleur de remplissage (argument '''fill''') et/ou la couleur de la bordure (argument '''colour''') : | ||
<pre> | <pre> | ||
qplot(Sepal.Length,data=iris,geom="histogram",fill=I("orange")) ## couleur de remplissage en orange | qplot(Sepal.Length,data=iris,geom="histogram",fill=I("orange")) ## couleur de remplissage en orange | ||
qplot(Sepal.Length,data=iris,geom="histogram",fill=I("orange"),colour=I("black")) ## et bordure en noir | qplot(Sepal.Length,data=iris,geom="histogram",fill=I("orange"),colour=I("black")) ## et bordure en noir | ||
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:histo_orange_qplot.png]] | ||
| + | |||
| + | [[Fichier:histo_orange_black_qplot.png]] | ||
De plus, QQPLOT permet d’adapter la largeur des classes utilisées pour tracer l’histogramme, et donc d’avoir un degré de précision plus ou moins élevé, grâce à l’argument binwidth. | De plus, QQPLOT permet d’adapter la largeur des classes utilisées pour tracer l’histogramme, et donc d’avoir un degré de précision plus ou moins élevé, grâce à l’argument binwidth. | ||
| Ligne 102 : | Ligne 112 : | ||
qplot(Sepal.Length,data=iris,geom="histogram",binwidth=0.05) ## classes ressérées | qplot(Sepal.Length,data=iris,geom="histogram",binwidth=0.05) ## classes ressérées | ||
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:histo_large_qplot.png]] | ||
| + | [[Fichier:histo_serre_qplot.png]] | ||
===== Courbe de densité ===== | ===== Courbe de densité ===== | ||
| Ligne 107 : | Ligne 120 : | ||
qplot(Sepal.Length,data=iris,geom="density",colour=I("red")) | qplot(Sepal.Length,data=iris,geom="density",colour=I("red")) | ||
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:courbe_densité_qplot.png]] | ||
| + | |||
| + | Il peut être intéressant de la combiner avec l'histogramme. | ||
===== Polynôme de fréquence ===== | ===== Polynôme de fréquence ===== | ||
| Ligne 112 : | Ligne 129 : | ||
qplot(Sepal.Length,data=iris,geom="freqpoly",colour=I("blue")) | qplot(Sepal.Length,data=iris,geom="freqpoly",colour=I("blue")) | ||
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:polyfreq_qplot.png]] | ||
===== Variable discrète ===== | ===== Variable discrète ===== | ||
| Ligne 119 : | Ligne 138 : | ||
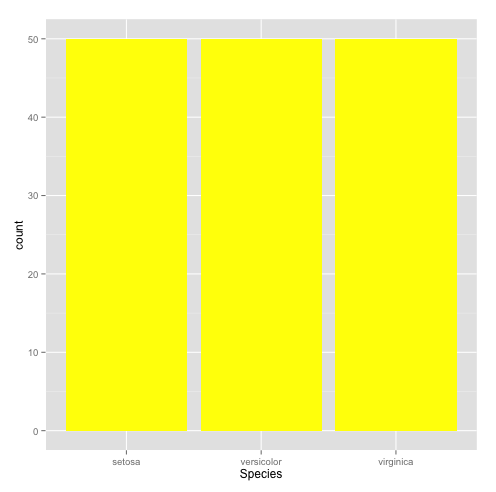
qplot(Species,data=iris,geom="bar",fill=I("yellow")) #par défaut | qplot(Species,data=iris,geom="bar",fill=I("yellow")) #par défaut | ||
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:barplot_qplot.png]] | ||
=== Deux dimensions === | === Deux dimensions === | ||
| − | ==== Deux variables continues | + | ==== Deux variables continues ==== |
===== Scatterplot ===== | ===== Scatterplot ===== | ||
| Ligne 131 : | Ligne 152 : | ||
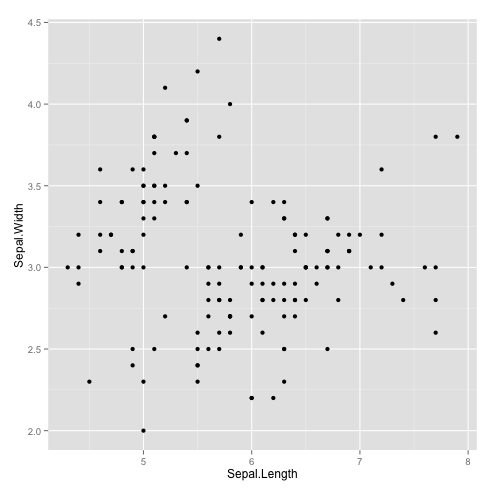
qplot(Sepal.Length,Sepal.Width, data=iris, geom="point") #par défaut | qplot(Sepal.Length,Sepal.Width, data=iris, geom="point") #par défaut | ||
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:scatt_base_qplot.png]] | ||
Les arguments de la fonction peuvent être modifiés, par exemple pour produire un graphique avec des points bleu (et non noirs).... | Les arguments de la fonction peuvent être modifiés, par exemple pour produire un graphique avec des points bleu (et non noirs).... | ||
| Ligne 137 : | Ligne 160 : | ||
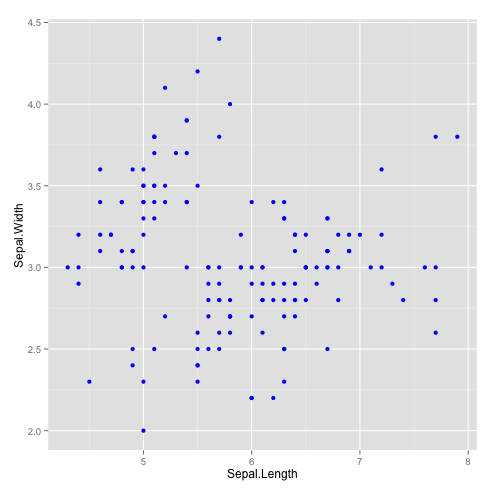
qplot(Sepal.Length,Sepal.Width, data=iris,colour = I("blue")) | qplot(Sepal.Length,Sepal.Width, data=iris,colour = I("blue")) | ||
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:scatt_bleu_qplot.png]] | ||
... ou pour produire un graphique avec des « + » à la place des points. | ... ou pour produire un graphique avec des « + » à la place des points. | ||
| Ligne 143 : | Ligne 168 : | ||
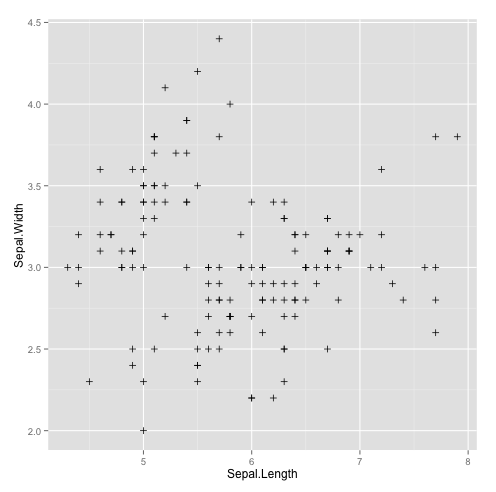
qplot(Sepal.Length,Sepal.Width, data=iris,shape=I(3)) | qplot(Sepal.Length,Sepal.Width, data=iris,shape=I(3)) | ||
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:scatt_plus_qplot.png]] | ||
Il est aussi possible de modifier la taille des points sur le graphique, à l’aide de l’argument size par exemple pour affiner les points ou les grossir(par défaut, size=I(2)). | Il est aussi possible de modifier la taille des points sur le graphique, à l’aide de l’argument size par exemple pour affiner les points ou les grossir(par défaut, size=I(2)). | ||
<pre> | <pre> | ||
## taille | ## taille | ||
| − | qplot(Sepal.Length,Sepal.Width, data=iris,size=I( | + | qplot(Sepal.Length,Sepal.Width, data=iris,size=I(3)) |
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:scatt_taille_qplot.png]] | ||
Enfin, l’argument alpha est utile en cas de nombre conséquent d’observations qui vont se superposer sur le graphique (ce qui n’est pas le cas ici). En ajustant la transparence, il permet d’améliorer la lisibilité du graphique. Par défaut alpha=I(1), et l’utilisateur peut fixer alpha=I(1/x), avec x le nombre nécessaire d’observations qui se superposent pour obtenir une couleur opaque. | Enfin, l’argument alpha est utile en cas de nombre conséquent d’observations qui vont se superposer sur le graphique (ce qui n’est pas le cas ici). En ajustant la transparence, il permet d’améliorer la lisibilité du graphique. Par défaut alpha=I(1), et l’utilisateur peut fixer alpha=I(1/x), avec x le nombre nécessaire d’observations qui se superposent pour obtenir une couleur opaque. | ||
| Ligne 155 : | Ligne 184 : | ||
qplot(Sepal.Length,Sepal.Width, data=iris,alpha=I(1/10)) | qplot(Sepal.Length,Sepal.Width, data=iris,alpha=I(1/10)) | ||
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:scatt_transparence_qplot.png]] | ||
===== Courbe de tendance ===== | ===== Courbe de tendance ===== | ||
| Ligne 160 : | Ligne 191 : | ||
qplot(Sepal.Length,Sepal.Width, data=iris, geom="smooth") | qplot(Sepal.Length,Sepal.Width, data=iris, geom="smooth") | ||
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:courbe_tendance_qplot.png]] | ||
à noter que la méthode pour tracer la courbe de tendance peut être changée par l’utilisateur grâce à l’argument «method= (par défaut, method=« loess » pour un nombre d’observations faible). Par exemple, method=« lm » permet de tracer une droite de regression linéaire. | à noter que la méthode pour tracer la courbe de tendance peut être changée par l’utilisateur grâce à l’argument «method= (par défaut, method=« loess » pour un nombre d’observations faible). Par exemple, method=« lm » permet de tracer une droite de regression linéaire. | ||
| Ligne 165 : | Ligne 198 : | ||
qplot(Sepal.Length,Sepal.Width, data=iris, geom="smooth",method="lm") # lm | qplot(Sepal.Length,Sepal.Width, data=iris, geom="smooth",method="lm") # lm | ||
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:courbelm_qplot.png]] | ||
Il peut être utile de combiner l'objet géométrique courbe de tendance avec un nuage de points : | Il peut être utile de combiner l'objet géométrique courbe de tendance avec un nuage de points : | ||
| Ligne 170 : | Ligne 205 : | ||
qplot(Sepal.Length,Sepal.Width, data=iris, geom=c("point","smooth"),method="lm") #combiné | qplot(Sepal.Length,Sepal.Width, data=iris, geom=c("point","smooth"),method="lm") #combiné | ||
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:scatt_lm_qplot.png]] | ||
Le tracé peut être plus ou moins sinueux grâce à l’argument span, allant de 0 à 1. | Le tracé peut être plus ou moins sinueux grâce à l’argument span, allant de 0 à 1. | ||
| Ligne 178 : | Ligne 215 : | ||
</pre> | </pre> | ||
| + | [[Fichier:scatt_lm_span1_qplot.png]] | ||
| + | [[Fichier:scatt_lm_span2_qplot.png]] | ||
| − | + | ==== Une variable continue, une discrète ==== | |
| − | + | Il n'est pas rare de vouloir représenter la distribution une variable continue, selon différentes sous-populations définies par une variable discrète. Pour cela on peut utiliser un boxplot. (voir [[Statistiques descriptives avec R]]). QPLOT va également permettre des représentations plus perfectionnées des graphiques présentés précédemment, en tenant compte de ces différentes sous-populations. | |
| + | |||
| + | ===== Boxplot ===== | ||
| + | <pre> | ||
#Une continue, une discrete | #Une continue, une discrete | ||
| − | qplot(Species,Sepal.Length, data=iris, geom="boxplot") | + | qplot(Species,Sepal.Length, data=iris, geom="boxplot",fill=I("purple")) |
| + | </pre> | ||
| − | + | [[Fichier:boxplot_qplot.png]] | |
| − | + | ===== Sous-populations : sur un même graphique ===== | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
On peut présenter les observations issues de différentes sous-populations définies par les niveaux de la variable facteur group, soit d’une couleur différente (contour ou remplissage), soit d’un symbole différent. | On peut présenter les observations issues de différentes sous-populations définies par les niveaux de la variable facteur group, soit d’une couleur différente (contour ou remplissage), soit d’un symbole différent. | ||
Rappel : | Rappel : | ||
| Ligne 201 : | Ligne 236 : | ||
* argument fill = remplissage | * argument fill = remplissage | ||
* argument shape = symbole | * argument shape = symbole | ||
| + | |||
Par exemple l’argument colour=group permet de présenter d'une couleur différente sur le nuage de points, les observations issues de différentes sous-populations, définies par les niveaux de la variable facteur group. | Par exemple l’argument colour=group permet de présenter d'une couleur différente sur le nuage de points, les observations issues de différentes sous-populations, définies par les niveaux de la variable facteur group. | ||
| Ligne 207 : | Ligne 243 : | ||
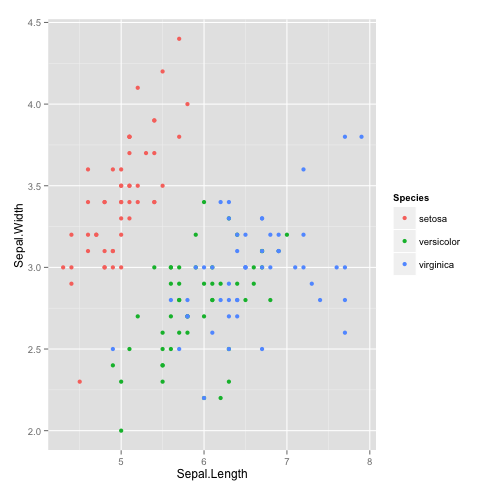
qplot(Sepal.Length,Sepal.Width, data=iris,colour=Species) | qplot(Sepal.Length,Sepal.Width, data=iris,colour=Species) | ||
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:scatt_Species_qplot.png]] | ||
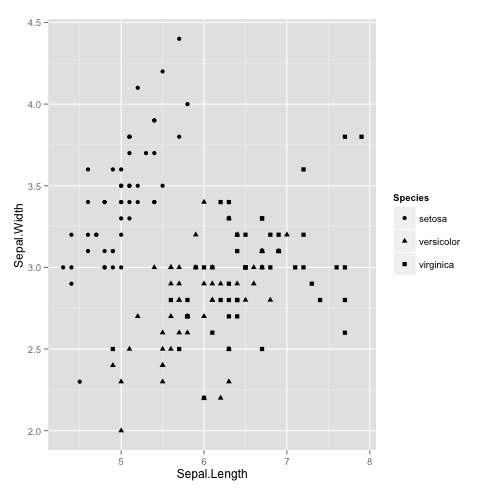
On peut faire de même sur un graphique de type scatterplot grâce à l’argument shape=group, avec cette fois des symboles différents, selon le niveau de la variable group. | On peut faire de même sur un graphique de type scatterplot grâce à l’argument shape=group, avec cette fois des symboles différents, selon le niveau de la variable group. | ||
| Ligne 212 : | Ligne 250 : | ||
qplot(Sepal.Length,Sepal.Width, data=iris,geom="point",shape=Species) #symboles | qplot(Sepal.Length,Sepal.Width, data=iris,geom="point",shape=Species) #symboles | ||
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:scatt_Species_symbole_qplot.png]] | ||
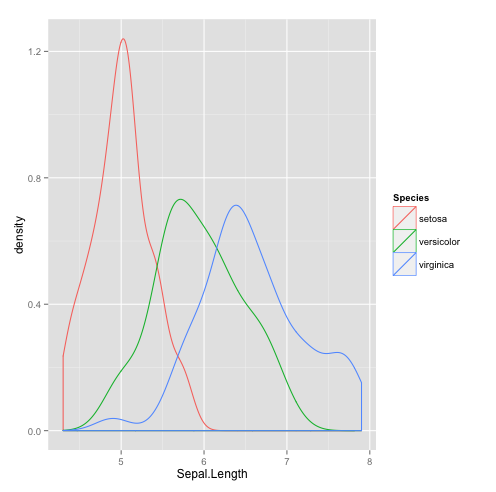
C’est aussi réalisable sur des courbes de densité (argument colour). | C’est aussi réalisable sur des courbes de densité (argument colour). | ||
| Ligne 218 : | Ligne 258 : | ||
qplot(Sepal.Length, data=iris,geom="density",colour=Species) | qplot(Sepal.Length, data=iris,geom="density",colour=Species) | ||
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:densité_Species_qplot.png]] | ||
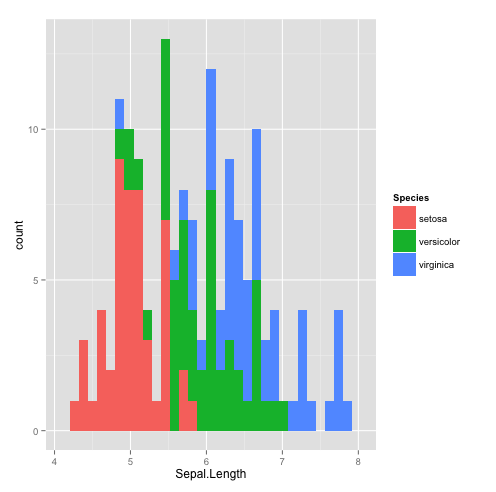
Pour un histogramme, l’agument à utiliser est fill=group pour modifier le remplissage. | Pour un histogramme, l’agument à utiliser est fill=group pour modifier le remplissage. | ||
| Ligne 225 : | Ligne 267 : | ||
</pre> | </pre> | ||
| − | ==== | + | [[Fichier:Histo_Species_qplot.png]] |
| + | |||
| + | |||
| + | Attention à ne pas confondre fill=I("red") (utilisé pour paramétrer le remplissage en rouge, valable aussi pour l'argument colour..) et fill=group (utilisé pour changer la couleur selon les sous-populations définies pas la variable group). Déclarer fill="red" et non fill=I("red") aurait pour conséquence de créer une nouvelle variable "red", et de représenter les observations de couleurs différentes selon les modalités prises par cette variable (soit une seule). R ne produira pas de message d'erreur, mais le graphique sera obtenu de manière erronée. | ||
| + | <pre> | ||
| + | #histogramme | ||
| + | qplot(Sepal.Length,data=iris,geom="histogram",fill="red") | ||
| + | </pre> | ||
| + | |||
| + | [[Fichier:Histo_faux_qplot.png]] | ||
| + | |||
| + | Le graphique correct serait : | ||
| + | <pre> | ||
| + | #histogramme | ||
| + | qplot(Sepal.Length,data=iris,geom="histogram",fill=I("red")) | ||
| + | </pre> | ||
| + | |||
| + | [[Fichier:Histo_faux_correct_qplot.png]] | ||
| + | |||
| + | |||
| + | ===== Sous-populations : différents graphiques ===== | ||
| + | |||
QPLOT offre aussi la possibilité de créer plusieurs graphiques juxtaposés, correspondant à un même graphique pour chacune des différentes sous-populations définies par une variable group. On utilise l’argument facets=. <br /> | QPLOT offre aussi la possibilité de créer plusieurs graphiques juxtaposés, correspondant à un même graphique pour chacune des différentes sous-populations définies par une variable group. On utilise l’argument facets=. <br /> | ||
| Ligne 232 : | Ligne 295 : | ||
Par exemple ici, l’histogramme pour la variable Sepal.Length, pour chaque espèce. On constate qu’il est préférable de les présenter verticalement, sinon les histogrammes sont écrasés et peu lisibles. | Par exemple ici, l’histogramme pour la variable Sepal.Length, pour chaque espèce. On constate qu’il est préférable de les présenter verticalement, sinon les histogrammes sont écrasés et peu lisibles. | ||
<pre> | <pre> | ||
| − | qplot(Sepal.Length,data=iris,facets = Species ~ .,geom="histogram",binwidth=0.1) #ordre vertical | + | qplot(Sepal.Length,data=iris,facets = Species ~ .,geom="histogram",binwidth=0.1,fill=I("orange")) #ordre vertical |
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:facet_vertical_qplot.png]] | ||
| + | |||
<pre> | <pre> | ||
| − | qplot(Sepal.Length,data=iris,facets = . ~ Species,geom="histogram",binwidth=0.1) #ordre horizontal | + | qplot(Sepal.Length,data=iris,facets = . ~ Species,geom="histogram",binwidth=0.1,fill=I("orange")) #ordre horizontal |
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:facet_horizontal_qplot.png]] | ||
==== Données en séries temporelles ==== | ==== Données en séries temporelles ==== | ||
| Ligne 249 : | Ligne 317 : | ||
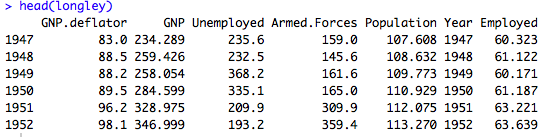
head(longley) | head(longley) | ||
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:head_longley.png]] | ||
==== Geom line ==== | ==== Geom line ==== | ||
| Ligne 256 : | Ligne 326 : | ||
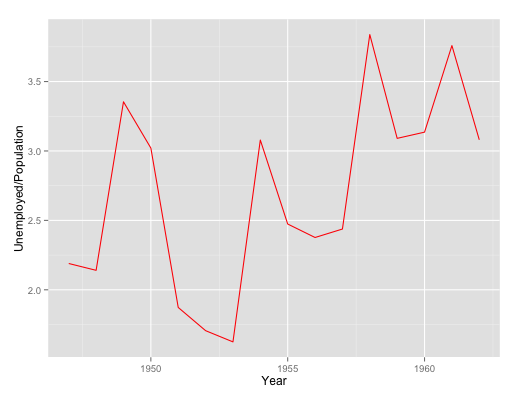
qplot(Year,Unemployed/Population, data=longley, geom="line",colour=I("red")) | qplot(Year,Unemployed/Population, data=longley, geom="line",colour=I("red")) | ||
</pre> | </pre> | ||
| + | |||
| + | [[Fichier:line_qplot.png]] | ||
==== Geom path ==== | ==== Geom path ==== | ||
| Ligne 264 : | Ligne 336 : | ||
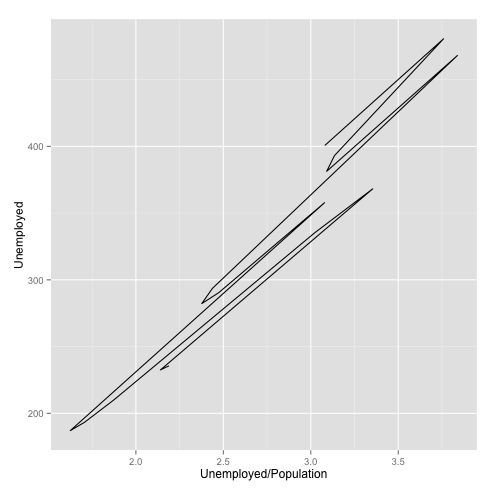
</pre> | </pre> | ||
| − | Ici, même sans ajouter la couleur le graphique est clair (il y a peu d’observations, et les tracés ne se superposent pas). Mais dans d'autres cas, l'ajout de ce dégradé de couleur selon l'année permet de rendre le graphique beaucoup plus lisible. | + | [[Fichier:path_qplot.png]] |
| + | |||
| + | Ici, même sans ajouter la couleur le graphique est clair (il y a peu d’observations, et les tracés ne se superposent pas). Mais dans d'autres cas, l'ajout de ce dégradé de couleur selon l'année permet de rendre le graphique beaucoup plus lisible. (Trois dimensions). | ||
<pre> | <pre> | ||
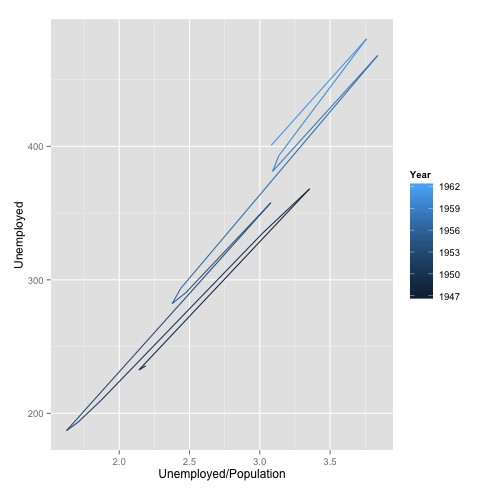
#Ajoute la couleur pour l'année | #Ajoute la couleur pour l'année | ||
| Ligne 270 : | Ligne 344 : | ||
</pre> | </pre> | ||
| + | [[Fichier:path_color_qplot.png]] | ||
| + | |||
| + | |||
| + | |||
| + | |||
| + | == GGPLOT == | ||
| + | |||
| + | ggplot est la seconde fonction du package ggplot2, et va permettre de produire des graphiques plus complexes et élaborés. | ||
| + | De plus, alors que la fonction qplot était relativement similaire dans sa syntaxe des fonctions traditionnelles permettant de tracer des graphiques sous R, la syntaxe utilisée par ggplot est totalement différente. | ||
| + | |||
| + | En effet, la fonction en elle-même n’a que deux arguments : | ||
| + | * data : les données (sous la forme data.frame) | ||
| + | * une fonction aes (pour "aesthetic mapping"), qui va contenir les variables et les paramètres utilisés pour l’esthétique du graphique. | ||
| + | |||
| + | ggplot fonctionne par l’ajout de calques (« layers »).<br /> | ||
| + | |||
| + | |||
| + | Ainsi, la commande suivante ne permet pas de tracer un graph, et nécessite pour cela d’ajouter au minimum un calque minimal. | ||
| + | <pre> | ||
| + | ggplot(data=iris,aes(Sepal.Length,Sepal.Width)) #no layers in plot | ||
| + | </pre> | ||
| + | Par exemple, si l’on souhaite tracer un nuage ce points, on ajoute le layer geom=« point » sous la forme suivante : ''ggplot(data=data,aes(x,y,….)) + layer(geom=« point »,……)''. C'est pourquoi, il est intéressant de "stocker" le graphique dans un objet R, et de le modifier par l'ajout successif de layers, comme ci-dessous. | ||
| + | <pre> | ||
| + | # Ajoute un layer minimal | ||
| + | plot<-ggplot(data=iris,aes(Sepal.Length,Sepal.Width)) | ||
| + | plot+layer(geom="point") | ||
| + | </pre> | ||
| + | |||
| + | === Layer === | ||
| + | Layer peut prendre différents arguments (le principal étant geom), par exemple : | ||
| + | * '''geom''' permet de spécifier le type de graphique souhaité (de manière similaire à l’usage dans la fonction qplot) | ||
| + | * '''geom_params''' va contenir tous les paramètres associés, permettant de personnaliser le graphique. | ||
| + | * '''stat''' consiste en une transformation statistique des données. De nouvelles variables vont être créées, qui résument les variables d’une certaine manière. Lorsque geom est utilisé, cette transformation va être automatiquement réalisée (selon le type de graphique souhaité). Elle peut éventuellement être l’identité. | ||
| + | Par exemple, la transformation associée par défaut à l’histogramme est « bin », et va créer trois nouvelles variables : count (le nombre d’observation dans chaque classe), density (le pourcentage divisé par la largeur de la classe), et x (le centre de la classe). | ||
| + | * '''stat_params''' contient les paramètres associés à cette transformation. Par exemple lorsque que l’on souhaite spécifier la largeur des classes utilisées pour l’histogramme, il va s’agir d’un paramètre stat, et non geom.<br /> | ||
| + | |||
| + | |||
| + | Ainsi, la commande ci-dessous va permettre de tracer un histogramme pour la longueur de la sépale, de couleur orange, avec une longueur de classe de 0.3. | ||
| + | <pre> | ||
| + | #Histogramme | ||
| + | ggplot(data=iris,aes(x=Sepal.Length)) + layer(geom="histogram", | ||
| + | geom_params=list(fill="orange"), | ||
| + | stat="bin", | ||
| + | stat_params=list(binwidth=2)) | ||
| + | </pre> | ||
| + | Cependant, cette syntaxe est relativement lourde, et peut être considérablement allégée. | ||
| + | |||
| + | Tout d’abord, l’argument layer(geom="histogram",geom_params=list(fill="orange") peut être remplacé par l’argument geom_histogram(fill=« orange »). De plus, en tenant compte du fait que chaque type de graphique (geom) est lié à une transformation statistique (stats), on peut finalement simplifier l’argument layer en : geom_histogram(fill="orange",binwidth=0.3) | ||
| + | <pre> | ||
| + | Ainsi, la commande suivante produit le même graphique. | ||
| + | #simplification | ||
| + | hist<-ggplot(data=iris,aes(x=Sepal.Length)) | ||
| + | hist <- hist + geom_histogram(fill="orange",binwidth=0.3) | ||
| + | hist | ||
| + | </pre> | ||
| + | NB : il est parfois nécessaire de demander à imprimer le graphique à l’écran.<br /> | ||
| + | |||
| + | |||
| + | |||
| + | On va raisonner de cette façon pour la suite. Ainsi les principaux layers pouvant être ajoutés vont être les suivants : | ||
| + | * geom_XXX() (par example : geom_point(), geom_histogram(), geom_vline(), etc.) : correspondant au type de graphique à créer | ||
| + | * stat_XXX() (par exemple : stat_bin(), stat_smooth(), etc.) : correspondant à la transformation statistique à utiliser | ||
| + | * scale_XXX() (par exemple : scale_x_continuous(), : contrôlant l’échelle utilisée pour les axes.
| ||
| + | * coord_XXX() : permettant de contrôler le système de coordonnées utilisé. | ||
| + | * facet_XXX() : permettant de faire des graphiques pour des sous-groupes | ||
| + | * position_XXX() : | ||
| + | |||
| + | ==== coord ==== | ||
| + | coord() (système de coordonnées à utiliser) et scale() (échelles des axes) déterminent la position de l’argument géométrique geom. | ||
| + | |||
| + | Par défaut : coord_cartesian (coordonnées cartésiennes « classiques », x est représenté sur l’axe des abscisses (de gauche à droite), et y sur celui des ordonnées (de bas en haut) | ||
| + | * coord_flip : inverse l’axe des x et des y | ||
| + | * coord_equal : même échelle pour l’axe des x et celui des y (cartésiens) | ||
| + | * coord_polar : coordonnées polaires : | ||
| + | * préciser la variable qui représente theta (l’angle) | ||
| + | * coord_trans : permet de transformer « manuellement » les axes (nécessite de préciser la transformation voulue) | ||
| + | * coord_map :<br /> | ||
| + | |||
| + | Par exemple, on inverse l'axe des x et celui des y : | ||
| + | <pre> | ||
| + | ## coord flip : boxplot | ||
| + | plot <- ggplot(iris, aes(y=Sepal.Length, x=Species)) | ||
| + | plot<- plot + geom_boxplot(fill="#FF9999") | ||
| + | plot<- plot + coord_flip() | ||
| + | plot | ||
| + | </pre> | ||
| + | |||
| + | <pre> | ||
| + | ## coord flip : scatter + lm | ||
| + | plot <- ggplot(iris, aes(x=Sepal.Length, y=Species)) | ||
| + | plot<- plot + geom_point(colour="red") +geom_smooth(method="lm") | ||
| + | plot<- plot + coord_flip() | ||
| + | plot | ||
| + | </pre> | ||
| + | |||
| + | On utilise des coordonnées polaires, et non plus cartésiennes. | ||
| + | <pre> | ||
| + | ## coord_polar | ||
| + | plot <- ggplot(iris, aes(Sepal.Length, Sepal.Width)) | ||
| + | plot<- plot + geom_point(colour="red")+geom_smooth(method="lm") | ||
| + | plot<- plot + coord_polar() | ||
| + | plot | ||
| + | </pre> | ||
| + | |||
| + | On utilise une échelle logarithmique pour l'axe des ordonnées. | ||
| + | <pre> | ||
| + | ## coord_trans | ||
| + | plot <- ggplot(iris, aes(Sepal.Length, Sepal.Width)) | ||
| + | plot<- plot + geom_point(colour="red")+geom_smooth(method="lm") | ||
| + | plot<- plot + coord_trans(ytrans="log10") | ||
| + | plot | ||
| + | </pre> | ||
| + | ==== scale ==== | ||
| + | L'argument scale permet de contrôler les axes du graphique, par exemple : la transformation, les limites des axes, le pas utilisé, les labels, etc. | ||
| + | ===== Changer les labels (axe discret) ===== | ||
| + | On peut changer les labels de l'axe des x (discret). On ajoute également un titre à cet axe, ainsi qu'à l'axe des y. | ||
| + | <pre> | ||
| + | # changer labels | ||
| + | plot <- ggplot(iris, aes(y=Sepal.Length, x=Species)) | ||
| + | plot<- plot + geom_boxplot(fill="#FF9999") | ||
| + | plot<- plot + scale_x_discrete(breaks=c("setosa", "versicolor", "virginica"),labels = c("Espèce A","Espèce B","Espèce C"),name="Code espèce de l'iris") | ||
| + | + scale_y_continuous(name="Longueur de la Sépale") | ||
| + | plot | ||
| + | </pre> | ||
| + | ===== Changer le pas ===== | ||
| + | Ici, on change le pas de l'axe des y pour un pas tous les 0.25. | ||
| + | <pre> | ||
| + | # changer le pas | ||
| + | plot <- ggplot(iris, aes(Sepal.Length, Sepal.Width)) | ||
| + | plot<- plot + geom_point(colour="red") | ||
| + | plot<-plot + scale_y_continuous(breaks=seq(0, 10, 0.25)) | ||
| + | plot | ||
| + | </pre> | ||
| + | ===== Changer les bornes ===== | ||
| + | On change les limites de l'axe des y (limite entre 3 et 4). | ||
| + | <pre> | ||
| + | # changer les limites | ||
| + | plot <- ggplot(iris, aes(Sepal.Length, Sepal.Width)) | ||
| + | plot<- plot + geom_point(colour="red") | ||
| + | plot<-plot + scale_y_continuous(limits=c(3,4)) | ||
| + | plot | ||
| + | </pre> | ||
| − | + | Attention, pour des graphiques de type scatterplot, cela va simplement exclure du graphique les observations en dehors de l'intervalle. Cependant, pour un graphique de type boxplot, si l'objectif est de "zoomer", le résultat sera alors un boxplot erroné (car portant uniquement sur les observations DE l'intervalle). | |
| + | <pre> | ||
| + | # changer les limites : boxplot | ||
| + | plot <- ggplot(iris, aes(y=Sepal.Length, x=Species)) | ||
| + | plot<- plot + geom_boxplot(fill="#FF9999") | ||
| + | plot<-plot + scale_y_continuous(limits=c(4.5,6.5)) | ||
| + | plot | ||
| + | </pre> | ||
| + | Il convient alors d'utiliser coord_cartesian() pour définir les limites de l'axe en "zoomant". | ||
<pre> | <pre> | ||
| − | + | #zoom : boxplot | |
| − | + | plot <- ggplot(iris, aes(y=Sepal.Length, x=Species)) | |
| + | plot<- plot + geom_boxplot(fill="#FF9999") | ||
| + | plot<-plot + coord_cartesian(ylim=c(4.5,6.5)) | ||
| + | plot | ||
</pre> | </pre> | ||
| + | |||
| + | ==== position ==== | ||
| + | position permet d'ajuster la position de l'objet géométrique. Comme pour stat, une modalité est associée par défaut à chaque geom. Il n’est donc pas nécessaire de le spécifier, sauf si l’on souhaite utiliser un argument différent de la valeur par défaut. | ||
| + | |||
| + | Les ajustement de position disponibles sont : | ||
| + | * position_identity - default pour la pluspart des geoms. | ||
| + | * position_jitter - default pour geom_jitter | ||
| + | * position_dodge - default pour geom_boxplot | ||
| + | * position_stack - default pour geom_bar, geom_histogram et geom_area | ||
| + | * position_fill - utile pour geom_bar,geom_histogram et geom_area | ||
| + | |||
| + | Par exemple, il peut être intéressant d'adapter la position dans le cas de barplots ou d'histogrammes.<br /> | ||
| + | |||
| + | Par défaut position_stack : | ||
| + | <pre> | ||
| + | ### position | ||
| + | plot <- ggplot(iris, aes(Sepal.Length_cat)) | ||
| + | plot<- plot + geom_bar(aes(fill=Species)) | ||
| + | plot | ||
| + | </pre> | ||
| + | |||
| + | Pour des bâtons de même taille : | ||
| + | <pre> | ||
| + | plot <- ggplot(iris, aes(Sepal.Length_cat)) | ||
| + | plot<- plot + geom_bar(aes(fill=Species),position="fill") | ||
| + | plot | ||
| + | </pre> | ||
| + | |||
| + | Pour obtenir des bâtons séparés pour chacune des modalités : | ||
| + | <pre> | ||
| + | plot <- ggplot(iris, aes(Sepal.Length_cat)) | ||
| + | plot<- plot + geom_bar(aes(fill=Species),position="dodge") | ||
| + | plot | ||
| + | </pre> | ||
| + | |||
| + | Attention à réfléchir à la position la plus adaptée. Ici par exemple, le nombre d'observation pour l'espèce virginica et la modalité "petit" recouvre celles des deux autres espèces... | ||
| + | <pre> | ||
| + | plot <- ggplot(iris, aes(Sepal.Length_cat)) | ||
| + | plot<- plot + geom_bar(aes(fill=Species),position="identity") | ||
| + | plot | ||
| + | </pre> | ||
| + | |||
| + | ==== facet ==== | ||
| + | Comme précédemment, facet permet de produire des graphiques séparés selon les différents groupes. | ||
| + | <pre> | ||
| + | #Grouping : horizontal | ||
| + | plot <- ggplot(iris, aes(Sepal.Length, Sepal.Width)) | ||
| + | plot<- plot + geom_point(colour="red") | ||
| + | plot<- plot + facet_grid(. ~ Species) | ||
| + | plot | ||
| + | |||
| + | #Grouping : graphs verticaux | ||
| + | plot <- ggplot(iris, aes(Sepal.Length, Sepal.Width)) | ||
| + | plot<- plot + geom_point(colour="red") | ||
| + | plot<- plot +facet_grid(Species~.) | ||
| + | plot | ||
| + | </pre> | ||
| + | |||
| + | Il est aussi possible de séparer les observations selon deux variables (discrètes). | ||
| + | Par exemple, on crée une nouvelle variable discrète pour illustrer (car la base de données n'en contient qu'une seule). Celle-ci prend la valeur "petit" si la longueur de la Sépale est <=5, "moyen si elle est comprise entre 5 et 6 (6 inclus), et "grand" si elle est >6. | ||
| + | |||
| + | <pre> | ||
| + | ## On crée une nouvelle variable facteur : | ||
| + | iris$Sepal.Length_cat[iris$Sepal.Length<=5]<-"petit" | ||
| + | iris$Sepal.Length_cat[iris$Sepal.Length<=6 & iris$Sepal.Length>5]<-"moyen" | ||
| + | iris$Sepal.Length_cat[iris$Sepal.Length>=6]<-"grand" | ||
| + | |||
| + | #Grouping : deux variables | ||
| + | plot <- ggplot(iris, aes(Sepal.Length, Sepal.Width)) | ||
| + | plot<- plot + geom_point(colour="red") | ||
| + | plot<- plot + facet_grid(Sepal.Length_cat ~ Species) | ||
| + | plot | ||
| + | <pre> | ||
| + | |||
| + | === Données par groupes === | ||
| + | Pour afficher les observations issues de groupes différents (ici d'espèces différentes) de couleurs différentes, et de formes différentes, on utilise : | ||
| + | <pre> | ||
| + | ## scatterplot : couleurs et symboles différents pour chaque groupe | ||
| + | plot <- ggplot(iris, aes(Sepal.Length, Sepal.Width)) | ||
| + | plot<- plot + geom_point(aes(color = Species,shape = Species)) | ||
| + | plot | ||
| + | </pre> | ||
| + | |||
| + | Pour changer manuellement les couleurs : | ||
| + | <pre> | ||
| + | plot<- plot + scale_colour_manual(values = c("red", "orange", "purple")) | ||
| + | plot<-plot + scale_shape_manual(values = c(16, 17, 18)) | ||
| + | plot | ||
| + | </pre> | ||
| + | |||
| + | === Changer le jeu de données === | ||
| + | Une fois le graphique créé, il est possible de l'appliquer à un jeu de données différent (à condition que les variables correspondent). Par exemple ici, on créee trois bases de données, contenant les mêmes variables. Pour cela, on divise la base initiale selon l’espèce. | ||
| + | On crée un graphique de type scatter plot, affichant une droite de regression linéaire pour le sous-jeu de données setosa. L’utilisation de '''« %+% »''' va permettre de produire le même graphique, pour le sous-jeu de données versicolor. | ||
| + | <pre> | ||
| + | #Data | ||
| + | iris_setosa <- subset(iris, Species == "setosa") | ||
| + | iris_versi <- subset(iris, Species == "versicolor") | ||
| + | iris_virgin <- subset(iris, Species == "virginica") | ||
| + | |||
| + | plot<-ggplot(iris_setosa,aes(Sepal.Length,Sepal.Width))+geom_point(colour="red") | ||
| + | plot | ||
| + | |||
| + | plot %+% iris_versi | ||
| + | </pre> | ||
| + | |||
| + | |||
| + | Ajouter un titre | ||
| + | Modifier le thème | ||
| + | |||
| + | Hello. And Bye. | ||
Version actuelle datée du 12 juin 2018 à 14:16
Ggplot2 est un package de R qui permet de réaliser des graphiques plus perfectionnés, et au design plus moderne. Pour cela, il existe deux fonctions :
- QPLOT (pour "Quick Plot") qui est la fonction "de base" du package (mais qui offre de grandes possibilités). Syntaxe générale : qplot(x,y,data=database)
- GGPLOT va permettre de créer des graphiques extrêmement perfectionnés. Sa syntaxe diffère de la syntaxe habituelle : ggplot(data=data,aes=(x,y)) +layers. La fonction ggplot (argument data et aes pour "aesthetic mapping") va permettre de créer le graphique en lui même, et les layers vont permettre d'ajouter des paramètres modifiant la représentation.
##### On installe le package
install.packages("ggplot2")
Une fois installé, il suffit de charger le package, avant de vouloir l'utiliser.
### On charge le package library(ggplot2)
Sommaire
Jeu de données :
Pour illustrer, on utilise le jeu de données « iris » de R, qui contient 150 observations de 5 variables : 4 variables de type quantitatif, et une de type qualitatif (Species).
- Sepal.Length : longueur de la sépale
- Sepal.Width : largeur de la sépale Petal.Length : longueur du pétale
- Petal.Width : largeur du pétale
- Species : espèce.
### On charge les données data(iris)
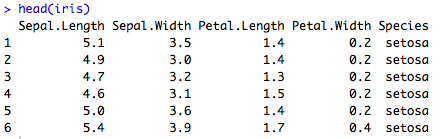
La fonction head(dataset) permet d’avoir un aperçu des premières lignes du jeu de données :
head(iris)
Attention ! pour utiliser ggplot2, les données doivent être sous la forme « data.frame », ce qui peut être vérifié par : class(dataset), et corrigé si besoin avec as.data.frame(dataset)
class(iris) as.data.frame(iris)
QPLOT
La syntaxe ressemble effectivement à celle de la fonction plot() (package "de base" de R).
qplot(x, y = NULL, ..., data, facets = NULL, margins = FALSE, geom = "auto", stat = list(NULL), position = list(NULL), xlim = c(NA,NA), ylim = c(NA, NA), log = "", main = NULL, xlab = deparse(substitute(x)), ylab = deparse(substitute(y)), asp = NA)
Objet géométrique
L’argument geom() (pour objet géométrique) va permettre de réaliser différents graphiques à une ou deux dimensions. Par défaut :
| Type de la variable | Objet par défaut |
|---|---|
| Quantitative | geom ="histogram" |
| Qualitative | geom="bar" |
| Deux variables quantitatives | geom="point" (scatterplot) |
De nombreux graphiques sont possibles (voir geom dans la documentation du package ggplot2, ou en page 56). Parmi les plus utilisés :
Pour une variable à analyser :
- histogram : histogramme (variable quantitative continue)
- density : trace la courbe de densité (variable quantitative continue)
- freqpoly : polynôme de fréquence (variable quantitative continue)
- bar : diagramme en bâton (variable discrète)
Pour deux variables :
- point : scatterplot (deux variables continues)
- smooth : trace une courbe de tendance et son écart-type (deux variables continues)
- boxplot : boxplot (une variable quantitative continue, une variable discrète)
Séries temporelles
- line : trace des lignes entre les points (de la gauche vers la droite)
- path : trace des lignes entre les points (dans n’importe quelle direction)
A noter également que les objets géométriques peuvent être combinés (à partir du moment où cela est raisonnable sur le plan statistique).
Une dimension
Variable quantitative, continue.
Histogramme
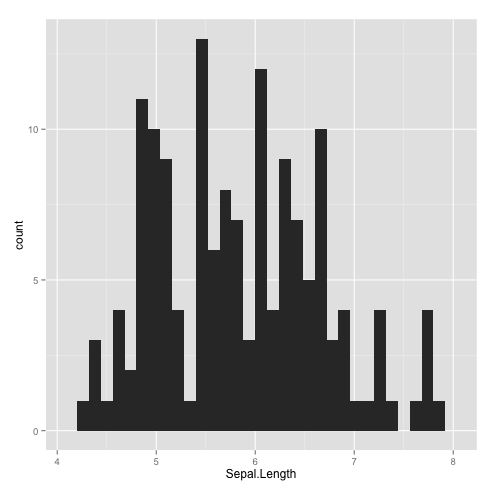
C'est le graphique tracé par défaut lorsque la variable indiquée est de type quantitatif (continu).
qplot(Sepal.Length,data=iris,geom="histogram") #par défaut
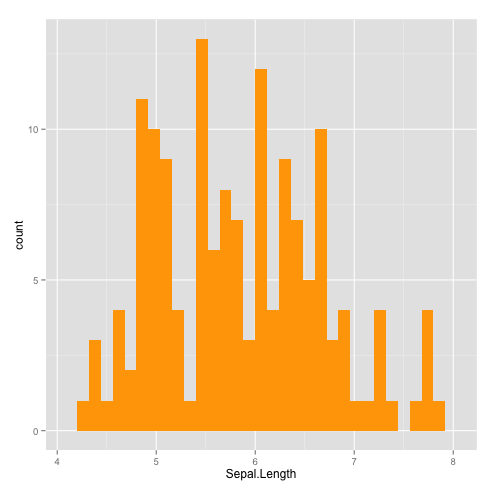
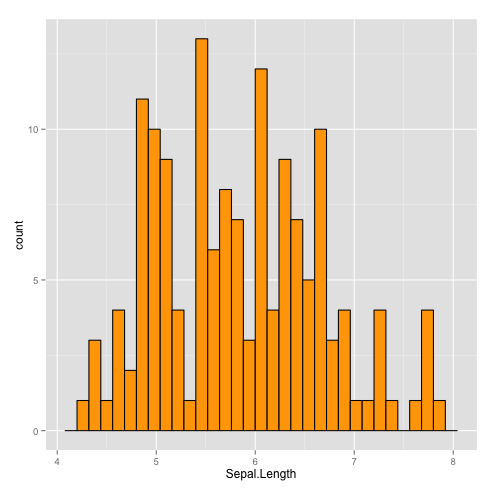
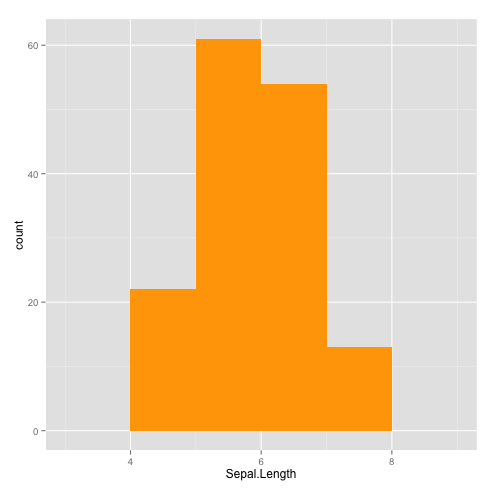
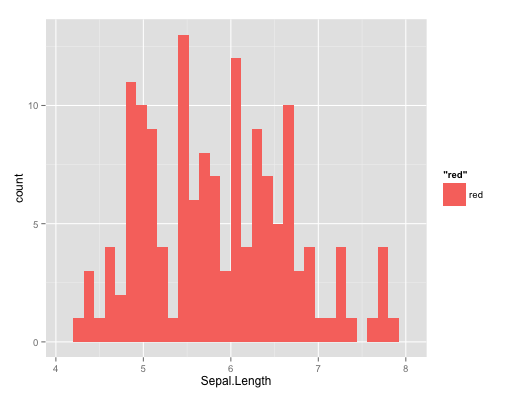
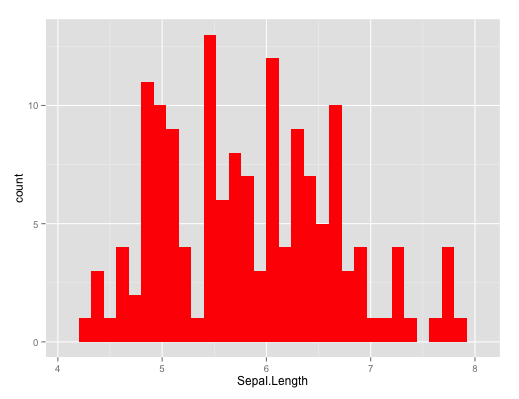
On peut l'améliorer : changer la couleur de remplissage (argument fill) et/ou la couleur de la bordure (argument colour) :
qplot(Sepal.Length,data=iris,geom="histogram",fill=I("orange")) ## couleur de remplissage en orange
qplot(Sepal.Length,data=iris,geom="histogram",fill=I("orange"),colour=I("black")) ## et bordure en noir
De plus, QQPLOT permet d’adapter la largeur des classes utilisées pour tracer l’histogramme, et donc d’avoir un degré de précision plus ou moins élevé, grâce à l’argument binwidth.
qplot(Sepal.Length,data=iris,geom="histogram",fill=I("orange"),binwidth=1) ## larges classes
qplot(Sepal.Length,data=iris,geom="histogram",binwidth=0.05) ## classes ressérées
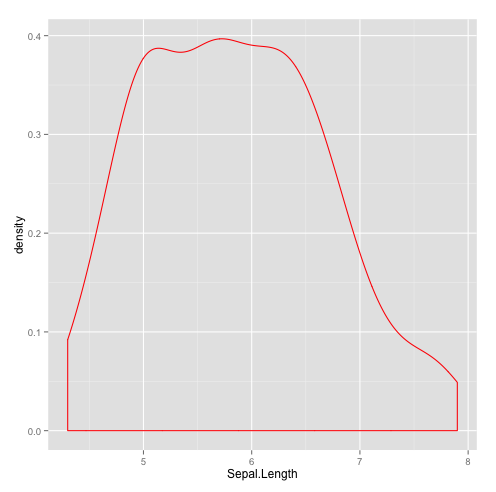
Courbe de densité
qplot(Sepal.Length,data=iris,geom="density",colour=I("red"))
Il peut être intéressant de la combiner avec l'histogramme.
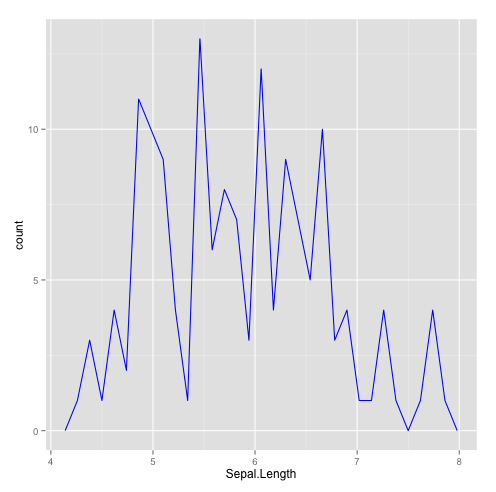
Polynôme de fréquence
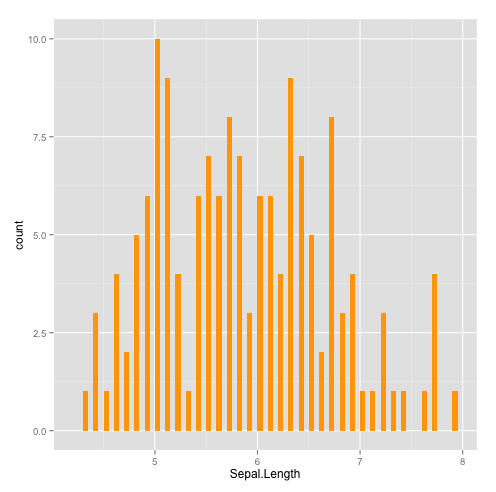
qplot(Sepal.Length,data=iris,geom="freqpoly",colour=I("blue"))
Variable discrète
Le graphique produit par défaut est un diagramme en bâton (à ne pas confondre avec un histogramme !!! voir Statistiques descriptives avec R)
qplot(Species,data=iris,geom="bar",fill=I("yellow")) #par défaut
Deux dimensions
Deux variables continues
Scatterplot
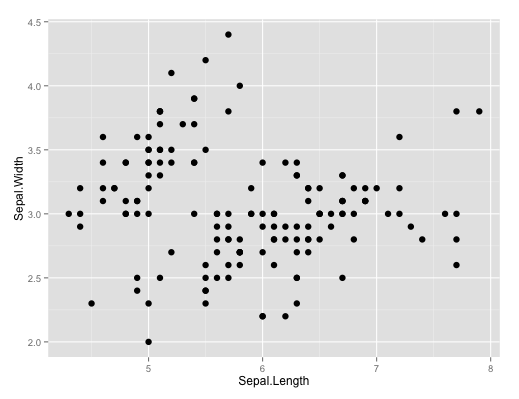
Le graphique produit par défaut par la fonction est un nuage de points classique.
#Deux variables continues qplot(Sepal.Length,Sepal.Width, data=iris, geom="point") #par défaut
Les arguments de la fonction peuvent être modifiés, par exemple pour produire un graphique avec des points bleu (et non noirs)....
## changer la couleur
qplot(Sepal.Length,Sepal.Width, data=iris,colour = I("blue"))
... ou pour produire un graphique avec des « + » à la place des points.
## changer le symbole qplot(Sepal.Length,Sepal.Width, data=iris,shape=I(3))
Il est aussi possible de modifier la taille des points sur le graphique, à l’aide de l’argument size par exemple pour affiner les points ou les grossir(par défaut, size=I(2)).
## taille qplot(Sepal.Length,Sepal.Width, data=iris,size=I(3))
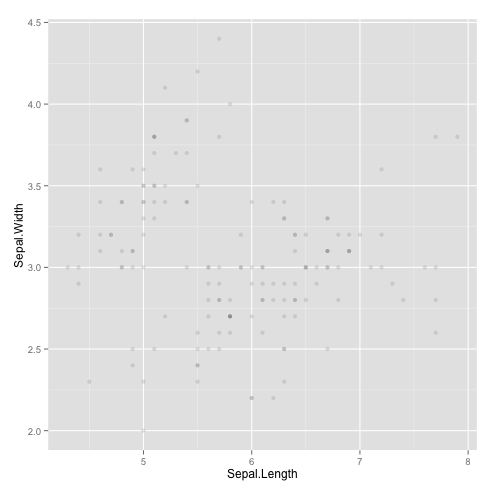
Enfin, l’argument alpha est utile en cas de nombre conséquent d’observations qui vont se superposer sur le graphique (ce qui n’est pas le cas ici). En ajustant la transparence, il permet d’améliorer la lisibilité du graphique. Par défaut alpha=I(1), et l’utilisateur peut fixer alpha=I(1/x), avec x le nombre nécessaire d’observations qui se superposent pour obtenir une couleur opaque.
## transparence qplot(Sepal.Length,Sepal.Width, data=iris,alpha=I(1/10))
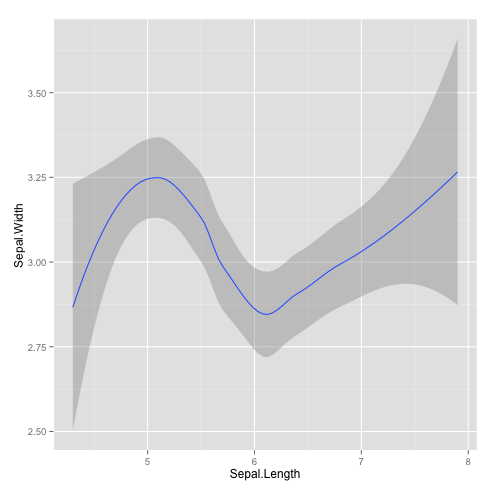
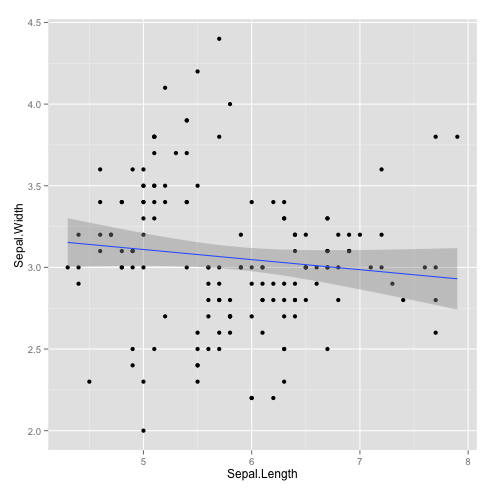
Courbe de tendance
qplot(Sepal.Length,Sepal.Width, data=iris, geom="smooth")
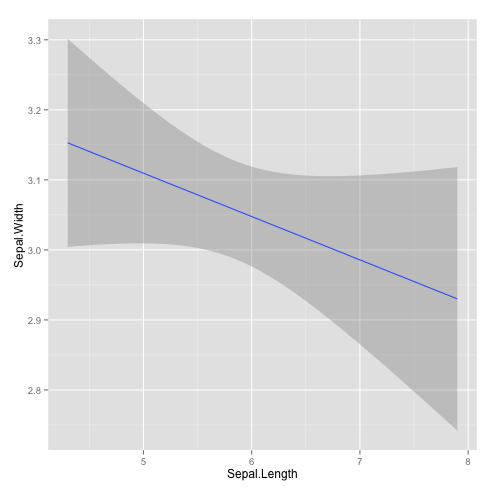
à noter que la méthode pour tracer la courbe de tendance peut être changée par l’utilisateur grâce à l’argument «method= (par défaut, method=« loess » pour un nombre d’observations faible). Par exemple, method=« lm » permet de tracer une droite de regression linéaire.
qplot(Sepal.Length,Sepal.Width, data=iris, geom="smooth",method="lm") # lm
Il peut être utile de combiner l'objet géométrique courbe de tendance avec un nuage de points :
qplot(Sepal.Length,Sepal.Width, data=iris, geom=c("point","smooth"),method="lm") #combiné
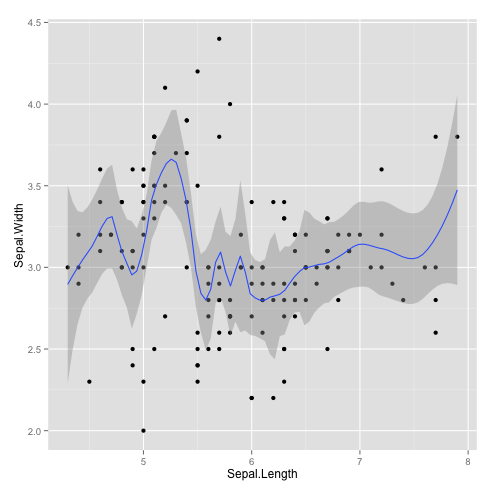
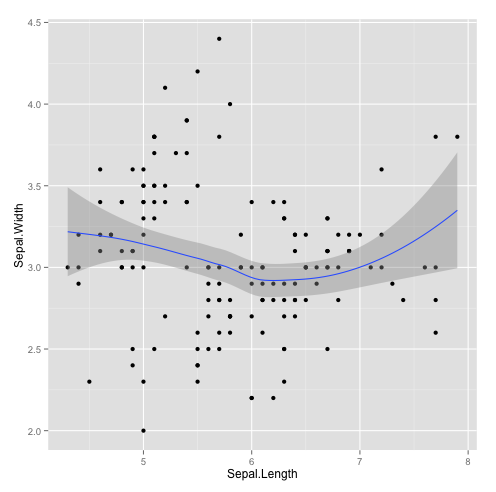
Le tracé peut être plus ou moins sinueux grâce à l’argument span, allant de 0 à 1.
##sinuosité
qplot(Sepal.Length,Sepal.Width, data=iris, geom=c("point","smooth"),span=0.2)
qplot(Sepal.Length,Sepal.Width, data=iris, geom=c("point","smooth"),span=1)
Une variable continue, une discrète
Il n'est pas rare de vouloir représenter la distribution une variable continue, selon différentes sous-populations définies par une variable discrète. Pour cela on peut utiliser un boxplot. (voir Statistiques descriptives avec R). QPLOT va également permettre des représentations plus perfectionnées des graphiques présentés précédemment, en tenant compte de ces différentes sous-populations.
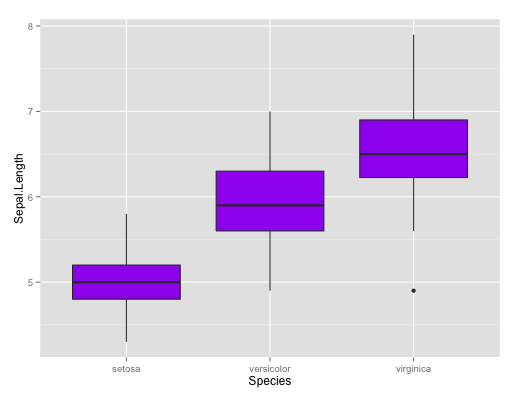
Boxplot
#Une continue, une discrete
qplot(Species,Sepal.Length, data=iris, geom="boxplot",fill=I("purple"))
Sous-populations : sur un même graphique
On peut présenter les observations issues de différentes sous-populations définies par les niveaux de la variable facteur group, soit d’une couleur différente (contour ou remplissage), soit d’un symbole différent. Rappel :
- argument colour = bordure
- argument fill = remplissage
- argument shape = symbole
Par exemple l’argument colour=group permet de présenter d'une couleur différente sur le nuage de points, les observations issues de différentes sous-populations, définies par les niveaux de la variable facteur group.
## ajoute de la couleur qplot(Sepal.Length,Sepal.Width, data=iris,colour=Species)
On peut faire de même sur un graphique de type scatterplot grâce à l’argument shape=group, avec cette fois des symboles différents, selon le niveau de la variable group.
qplot(Sepal.Length,Sepal.Width, data=iris,geom="point",shape=Species) #symboles
C’est aussi réalisable sur des courbes de densité (argument colour).
# densité qplot(Sepal.Length, data=iris,geom="density",colour=Species)
Pour un histogramme, l’agument à utiliser est fill=group pour modifier le remplissage.
#histogramme qplot(Sepal.Length,data=iris,geom="histogram",fill=Species)
Attention à ne pas confondre fill=I("red") (utilisé pour paramétrer le remplissage en rouge, valable aussi pour l'argument colour..) et fill=group (utilisé pour changer la couleur selon les sous-populations définies pas la variable group). Déclarer fill="red" et non fill=I("red") aurait pour conséquence de créer une nouvelle variable "red", et de représenter les observations de couleurs différentes selon les modalités prises par cette variable (soit une seule). R ne produira pas de message d'erreur, mais le graphique sera obtenu de manière erronée.
#histogramme qplot(Sepal.Length,data=iris,geom="histogram",fill="red")
Le graphique correct serait :
#histogramme
qplot(Sepal.Length,data=iris,geom="histogram",fill=I("red"))
Sous-populations : différents graphiques
QPLOT offre aussi la possibilité de créer plusieurs graphiques juxtaposés, correspondant à un même graphique pour chacune des différentes sous-populations définies par une variable group. On utilise l’argument facets=.
facets= . ~ group va présenter les graphiques cote à cote (horizontalement), tandis que facets= group ~ . les superposera (verticalement).
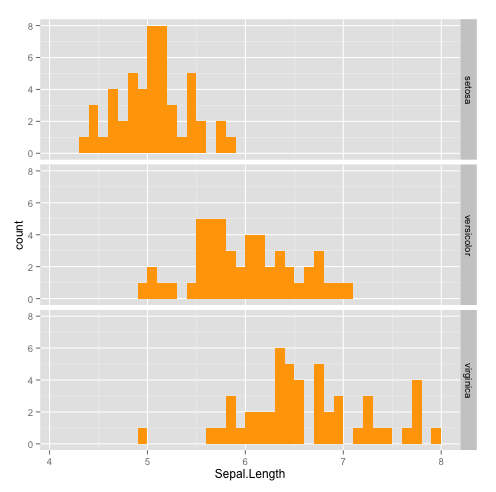
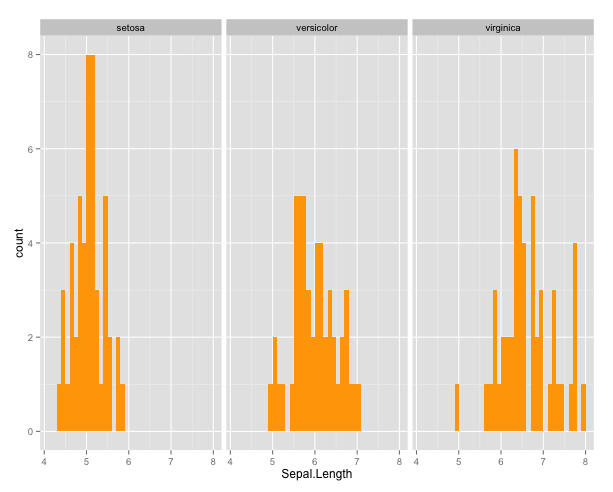
Par exemple ici, l’histogramme pour la variable Sepal.Length, pour chaque espèce. On constate qu’il est préférable de les présenter verticalement, sinon les histogrammes sont écrasés et peu lisibles.
qplot(Sepal.Length,data=iris,facets = Species ~ .,geom="histogram",binwidth=0.1,fill=I("orange")) #ordre vertical
qplot(Sepal.Length,data=iris,facets = . ~ Species,geom="histogram",binwidth=0.1,fill=I("orange")) #ordre horizontal
Données en séries temporelles
Line et path sont utilisés dans le cas de séries temporelles. On rappelle que la différence entre les deux provient du fait que path relie les points dans n’importe quelle direction (pourvu qu’il apparaissent en suivant dans la base de données), tandis que line relie les points de gauche à droite.
Ainsi, on utilisera line, par exemple pour tracer l’évolution d’un phénomène en fonction de la date, tandis que l’on utilisera path pour relier les différents points selon l’ordre chronologique, dans un graphique à deux dimensions (dont aucune n’est temporelle).
Pour illustrer, étant donné que les données "iris" sont en coupe transversale, on utilise le jeu de données « longley » qui contient des données macro-économiques annuelles (16 observations, entre 1947 et 1962).
data(longley) #on charge le jeu de données head(longley)
Geom line
Par exemple, on peut utiliser l’argument line pour tracer l’évolution temporelle du taux de chômage.
#evolution temporelle du nombre de personnes au chômage.
qplot(Year,Unemployed/Population, data=longley, geom="line",colour=I("red"))
Geom path
On trace ensuite par exemple le nombre de personnes au chômage en fonction du taux de chômage. L’argument path va permettre de relier les points pour visualiser la variation d’année en année.
#Nombre de personnes au chômage en fonction du taux de chômage qplot(Unemployed/Population,Unemployed, data=longley, geom="path")
Ici, même sans ajouter la couleur le graphique est clair (il y a peu d’observations, et les tracés ne se superposent pas). Mais dans d'autres cas, l'ajout de ce dégradé de couleur selon l'année permet de rendre le graphique beaucoup plus lisible. (Trois dimensions).
#Ajoute la couleur pour l'année qplot(Unemployed/Population,Unemployed, data=longley, geom="path",colour=Year)
GGPLOT
ggplot est la seconde fonction du package ggplot2, et va permettre de produire des graphiques plus complexes et élaborés. De plus, alors que la fonction qplot était relativement similaire dans sa syntaxe des fonctions traditionnelles permettant de tracer des graphiques sous R, la syntaxe utilisée par ggplot est totalement différente.
En effet, la fonction en elle-même n’a que deux arguments :
- data : les données (sous la forme data.frame)
- une fonction aes (pour "aesthetic mapping"), qui va contenir les variables et les paramètres utilisés pour l’esthétique du graphique.
ggplot fonctionne par l’ajout de calques (« layers »).
Ainsi, la commande suivante ne permet pas de tracer un graph, et nécessite pour cela d’ajouter au minimum un calque minimal.
ggplot(data=iris,aes(Sepal.Length,Sepal.Width)) #no layers in plot
Par exemple, si l’on souhaite tracer un nuage ce points, on ajoute le layer geom=« point » sous la forme suivante : ggplot(data=data,aes(x,y,….)) + layer(geom=« point »,……). C'est pourquoi, il est intéressant de "stocker" le graphique dans un objet R, et de le modifier par l'ajout successif de layers, comme ci-dessous.
# Ajoute un layer minimal plot<-ggplot(data=iris,aes(Sepal.Length,Sepal.Width)) plot+layer(geom="point")
Layer
Layer peut prendre différents arguments (le principal étant geom), par exemple :
- geom permet de spécifier le type de graphique souhaité (de manière similaire à l’usage dans la fonction qplot)
- geom_params va contenir tous les paramètres associés, permettant de personnaliser le graphique.
- stat consiste en une transformation statistique des données. De nouvelles variables vont être créées, qui résument les variables d’une certaine manière. Lorsque geom est utilisé, cette transformation va être automatiquement réalisée (selon le type de graphique souhaité). Elle peut éventuellement être l’identité.
Par exemple, la transformation associée par défaut à l’histogramme est « bin », et va créer trois nouvelles variables : count (le nombre d’observation dans chaque classe), density (le pourcentage divisé par la largeur de la classe), et x (le centre de la classe).
- stat_params contient les paramètres associés à cette transformation. Par exemple lorsque que l’on souhaite spécifier la largeur des classes utilisées pour l’histogramme, il va s’agir d’un paramètre stat, et non geom.
Ainsi, la commande ci-dessous va permettre de tracer un histogramme pour la longueur de la sépale, de couleur orange, avec une longueur de classe de 0.3.
#Histogramme
ggplot(data=iris,aes(x=Sepal.Length)) + layer(geom="histogram",
geom_params=list(fill="orange"),
stat="bin",
stat_params=list(binwidth=2))
Cependant, cette syntaxe est relativement lourde, et peut être considérablement allégée.
Tout d’abord, l’argument layer(geom="histogram",geom_params=list(fill="orange") peut être remplacé par l’argument geom_histogram(fill=« orange »). De plus, en tenant compte du fait que chaque type de graphique (geom) est lié à une transformation statistique (stats), on peut finalement simplifier l’argument layer en : geom_histogram(fill="orange",binwidth=0.3)
Ainsi, la commande suivante produit le même graphique. #simplification hist<-ggplot(data=iris,aes(x=Sepal.Length)) hist <- hist + geom_histogram(fill="orange",binwidth=0.3) hist
NB : il est parfois nécessaire de demander à imprimer le graphique à l’écran.
On va raisonner de cette façon pour la suite. Ainsi les principaux layers pouvant être ajoutés vont être les suivants :
- geom_XXX() (par example : geom_point(), geom_histogram(), geom_vline(), etc.) : correspondant au type de graphique à créer
- stat_XXX() (par exemple : stat_bin(), stat_smooth(), etc.) : correspondant à la transformation statistique à utiliser
- scale_XXX() (par exemple : scale_x_continuous(), : contrôlant l’échelle utilisée pour les axes.
- coord_XXX() : permettant de contrôler le système de coordonnées utilisé.
- facet_XXX() : permettant de faire des graphiques pour des sous-groupes
- position_XXX() :
coord
coord() (système de coordonnées à utiliser) et scale() (échelles des axes) déterminent la position de l’argument géométrique geom.
Par défaut : coord_cartesian (coordonnées cartésiennes « classiques », x est représenté sur l’axe des abscisses (de gauche à droite), et y sur celui des ordonnées (de bas en haut)
- coord_flip : inverse l’axe des x et des y
- coord_equal : même échelle pour l’axe des x et celui des y (cartésiens)
- coord_polar : coordonnées polaires :
- préciser la variable qui représente theta (l’angle)
- coord_trans : permet de transformer « manuellement » les axes (nécessite de préciser la transformation voulue)
- coord_map :
Par exemple, on inverse l'axe des x et celui des y :
## coord flip : boxplot plot <- ggplot(iris, aes(y=Sepal.Length, x=Species)) plot<- plot + geom_boxplot(fill="#FF9999") plot<- plot + coord_flip() plot
## coord flip : scatter + lm plot <- ggplot(iris, aes(x=Sepal.Length, y=Species)) plot<- plot + geom_point(colour="red") +geom_smooth(method="lm") plot<- plot + coord_flip() plot
On utilise des coordonnées polaires, et non plus cartésiennes.
## coord_polar plot <- ggplot(iris, aes(Sepal.Length, Sepal.Width)) plot<- plot + geom_point(colour="red")+geom_smooth(method="lm") plot<- plot + coord_polar() plot
On utilise une échelle logarithmique pour l'axe des ordonnées.
## coord_trans plot <- ggplot(iris, aes(Sepal.Length, Sepal.Width)) plot<- plot + geom_point(colour="red")+geom_smooth(method="lm") plot<- plot + coord_trans(ytrans="log10") plot
scale
L'argument scale permet de contrôler les axes du graphique, par exemple : la transformation, les limites des axes, le pas utilisé, les labels, etc.
Changer les labels (axe discret)
On peut changer les labels de l'axe des x (discret). On ajoute également un titre à cet axe, ainsi qu'à l'axe des y.
# changer labels
plot <- ggplot(iris, aes(y=Sepal.Length, x=Species))
plot<- plot + geom_boxplot(fill="#FF9999")
plot<- plot + scale_x_discrete(breaks=c("setosa", "versicolor", "virginica"),labels = c("Espèce A","Espèce B","Espèce C"),name="Code espèce de l'iris")
+ scale_y_continuous(name="Longueur de la Sépale")
plot
Changer le pas
Ici, on change le pas de l'axe des y pour un pas tous les 0.25.
# changer le pas plot <- ggplot(iris, aes(Sepal.Length, Sepal.Width)) plot<- plot + geom_point(colour="red") plot<-plot + scale_y_continuous(breaks=seq(0, 10, 0.25)) plot
Changer les bornes
On change les limites de l'axe des y (limite entre 3 et 4).
# changer les limites plot <- ggplot(iris, aes(Sepal.Length, Sepal.Width)) plot<- plot + geom_point(colour="red") plot<-plot + scale_y_continuous(limits=c(3,4)) plot
Attention, pour des graphiques de type scatterplot, cela va simplement exclure du graphique les observations en dehors de l'intervalle. Cependant, pour un graphique de type boxplot, si l'objectif est de "zoomer", le résultat sera alors un boxplot erroné (car portant uniquement sur les observations DE l'intervalle).
# changer les limites : boxplot plot <- ggplot(iris, aes(y=Sepal.Length, x=Species)) plot<- plot + geom_boxplot(fill="#FF9999") plot<-plot + scale_y_continuous(limits=c(4.5,6.5)) plot
Il convient alors d'utiliser coord_cartesian() pour définir les limites de l'axe en "zoomant".
#zoom : boxplot plot <- ggplot(iris, aes(y=Sepal.Length, x=Species)) plot<- plot + geom_boxplot(fill="#FF9999") plot<-plot + coord_cartesian(ylim=c(4.5,6.5)) plot
position
position permet d'ajuster la position de l'objet géométrique. Comme pour stat, une modalité est associée par défaut à chaque geom. Il n’est donc pas nécessaire de le spécifier, sauf si l’on souhaite utiliser un argument différent de la valeur par défaut.
Les ajustement de position disponibles sont :
- position_identity - default pour la pluspart des geoms.
- position_jitter - default pour geom_jitter
- position_dodge - default pour geom_boxplot
- position_stack - default pour geom_bar, geom_histogram et geom_area
- position_fill - utile pour geom_bar,geom_histogram et geom_area
Par exemple, il peut être intéressant d'adapter la position dans le cas de barplots ou d'histogrammes.
Par défaut position_stack :
### position plot <- ggplot(iris, aes(Sepal.Length_cat)) plot<- plot + geom_bar(aes(fill=Species)) plot
Pour des bâtons de même taille :
plot <- ggplot(iris, aes(Sepal.Length_cat)) plot<- plot + geom_bar(aes(fill=Species),position="fill") plot
Pour obtenir des bâtons séparés pour chacune des modalités :
plot <- ggplot(iris, aes(Sepal.Length_cat)) plot<- plot + geom_bar(aes(fill=Species),position="dodge") plot
Attention à réfléchir à la position la plus adaptée. Ici par exemple, le nombre d'observation pour l'espèce virginica et la modalité "petit" recouvre celles des deux autres espèces...
plot <- ggplot(iris, aes(Sepal.Length_cat)) plot<- plot + geom_bar(aes(fill=Species),position="identity") plot
facet
Comme précédemment, facet permet de produire des graphiques séparés selon les différents groupes.
#Grouping : horizontal plot <- ggplot(iris, aes(Sepal.Length, Sepal.Width)) plot<- plot + geom_point(colour="red") plot<- plot + facet_grid(. ~ Species) plot #Grouping : graphs verticaux plot <- ggplot(iris, aes(Sepal.Length, Sepal.Width)) plot<- plot + geom_point(colour="red") plot<- plot +facet_grid(Species~.) plot
Il est aussi possible de séparer les observations selon deux variables (discrètes). Par exemple, on crée une nouvelle variable discrète pour illustrer (car la base de données n'en contient qu'une seule). Celle-ci prend la valeur "petit" si la longueur de la Sépale est <=5, "moyen si elle est comprise entre 5 et 6 (6 inclus), et "grand" si elle est >6.
## On crée une nouvelle variable facteur : iris$Sepal.Length_cat[iris$Sepal.Length<=5]<-"petit" iris$Sepal.Length_cat[iris$Sepal.Length<=6 & iris$Sepal.Length>5]<-"moyen" iris$Sepal.Length_cat[iris$Sepal.Length>=6]<-"grand" #Grouping : deux variables plot <- ggplot(iris, aes(Sepal.Length, Sepal.Width)) plot<- plot + geom_point(colour="red") plot<- plot + facet_grid(Sepal.Length_cat ~ Species) plot <pre> === Données par groupes === Pour afficher les observations issues de groupes différents (ici d'espèces différentes) de couleurs différentes, et de formes différentes, on utilise : <pre> ## scatterplot : couleurs et symboles différents pour chaque groupe plot <- ggplot(iris, aes(Sepal.Length, Sepal.Width)) plot<- plot + geom_point(aes(color = Species,shape = Species)) plot
Pour changer manuellement les couleurs :
plot<- plot + scale_colour_manual(values = c("red", "orange", "purple"))
plot<-plot + scale_shape_manual(values = c(16, 17, 18))
plot
Changer le jeu de données
Une fois le graphique créé, il est possible de l'appliquer à un jeu de données différent (à condition que les variables correspondent). Par exemple ici, on créee trois bases de données, contenant les mêmes variables. Pour cela, on divise la base initiale selon l’espèce. On crée un graphique de type scatter plot, affichant une droite de regression linéaire pour le sous-jeu de données setosa. L’utilisation de « %+% » va permettre de produire le même graphique, pour le sous-jeu de données versicolor.
#Data iris_setosa <- subset(iris, Species == "setosa") iris_versi <- subset(iris, Species == "versicolor") iris_virgin <- subset(iris, Species == "virginica") plot<-ggplot(iris_setosa,aes(Sepal.Length,Sepal.Width))+geom_point(colour="red") plot plot %+% iris_versi
Ajouter un titre
Modifier le thème
Hello. And Bye.